来源:yujie OxfordNanopore 时间:2018年7月11日 09:30
导读:
传染病预防控制国家重点实验室传染病诊断组利用纳米孔测序技术进行病原菌检测及基因组组装,获得了进展。Oxford Nanopore技术将以其测序速度更快、超长读长、现场操作性更强等优势将为未来传染病疫情现场控制模式带来技术革命。
近日,中国疾控中心传染病预防控制所、传染病预防控制国家重点实验室张建中研究员课题组利用牛津纳米孔测序技术(Oxford Nanopore Technology)在病原菌检测方面取得系列进展。
课题组尤元海针对当前我国流行的重要细菌性传染病--猩红热的病原体A族链球菌(Group A streptococcus, GAS)[1],利用Oxford Nanopore技术进行了基因组完成图的测定。

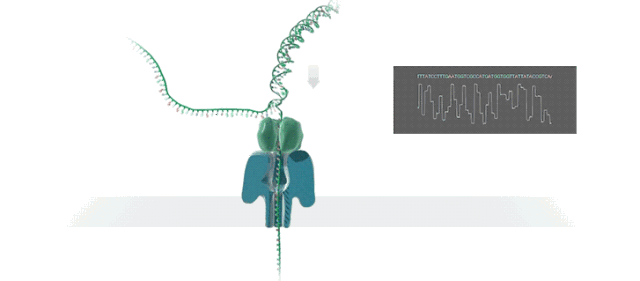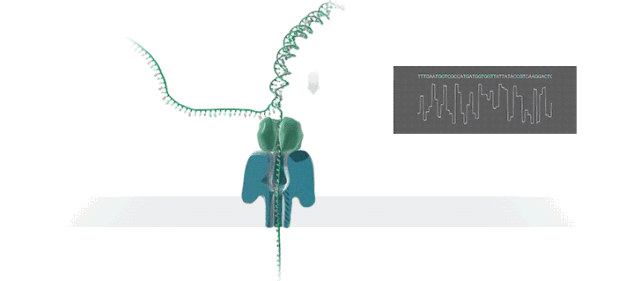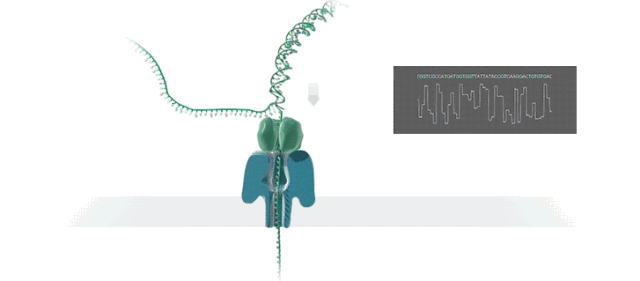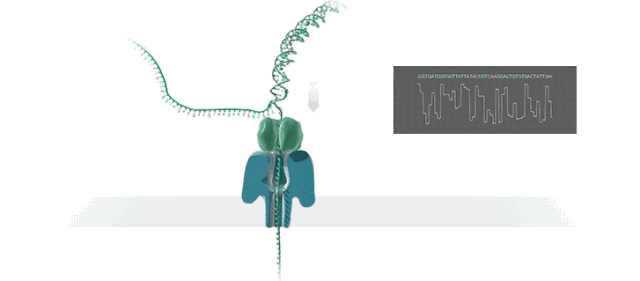
图1. MinION测序设备及测序原理示意图(引自https://nanoporetech.com)
课题组提取了自猩红热高发区分离的GAS菌株TJ11-001的基因组,利用纳米孔测序设备MinION、R9.4 Flowcell进行了测序。结果显示,Oxford Nanopore测序技术在10分钟内产生的reads数据即可即时获取GAS基因组中全部重要的耐药基因(红霉素耐药基因ermB、四环素耐药基因tetM等)、毒力基因(红疹毒素speC、链球菌超抗原ssa等),并确定了该菌株为emm12基因型。单独用Oxford Nanopore的长reads在1小时内即可组装达到GAS基因组平均长度(1.85Mb),准确率达到99%以上。同时可获得包括长度为61kb的ICE-HKU937和长度为46kb的prophage HKU.vir等在内的全部移动元件序列信息(图3),这些移动元件往往携带上述多种耐药基因和毒力基因在GAS不同血清型菌株间播散,可能是引起猩红热发病率上升的重要因素,对这些大片段基因组移动元件的监测对于我国乃至全球猩红热防控具有重要意义。
基于常规的快速基因组提取方法,本课题组所得nanopore测序数据最长read为184kb,通过优化提取方法将可获得更长读长。利用Oxford Nanopore与Illumina二代测序数据相结合进行组装可以使组装完成图的准确率达到99.9%以上。目前TJ11-001的基因组完成图及相关nanopore reads数据已在GenBank及SRA数据库共享。部分研究内容已在Genome Announcements发布[2]。
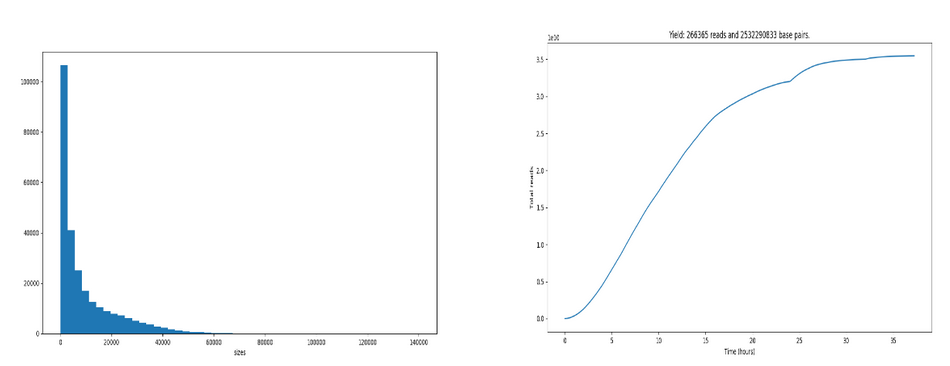
图2. Oxford Nanopore MinION 产生的reads长度及数据量分布
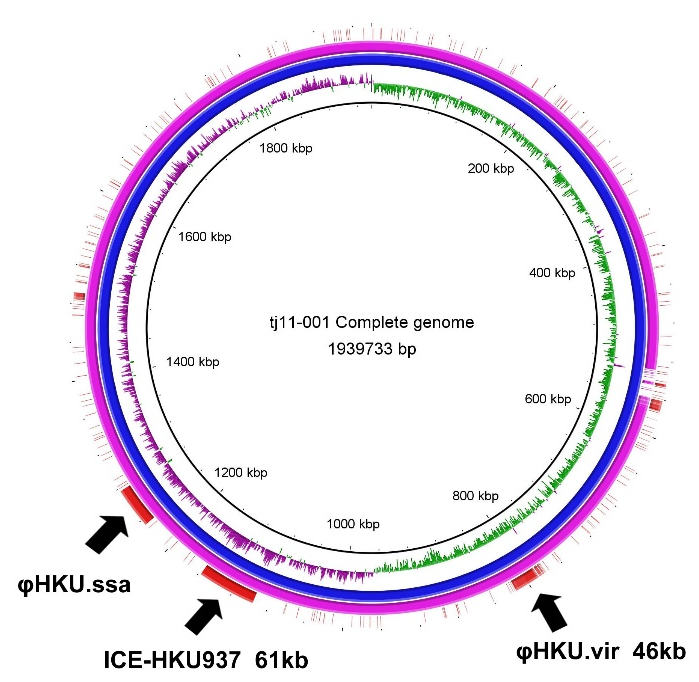
图3. 基于纳米孔测序组装结果快速鉴定GAS移动元件
为探索低成本多病原混合测序模式,课题组采用Barcoding方式同时测试了原核、真核等多物种混合建库模式,并成功完成相应物种的数据分拣及基因组拼接。Flowcell是Oxford Nanopore测序较昂贵的关键耗材,尽管Oxford Nanopore开发了针对重复利用Flowcell的Wash kit (清洗试剂盒),但根据本课题组的测试结果,在分析两种基因组长度接近的不同菌种时,经过清洗后再测序获得的数据中仍可残留30%以上前一种样品的reads,这需要用其参考基因组进行mapping、过滤后才能使用,因此,对于基因组高度相近的同一物种的不同菌株,采用这种清洗再生的方式进行测序可能无法准确进行mapping和过滤。这也是目前Oxford Nanopore技术在Flowcell重复利用方面有待改进的一个环节。
课题组与北京鑫汇普瑞科技发展有限公司合作,针对Oxford Nanopore实时数据分析的优势开发了一系列基于Windows操作系统、图形界面的快速自动化分析方案,使得从样本DNA提取到建库、基因分型及毒力与耐药等关键基因的读取,再到基因组完成图的组装,整个过程可在4-6小时内完成。该软件同时实现了从第一条nanopore read下线后的实时动态数据分析,实时显示每条read的种属分类、序列比对结果以及带有何种耐药、毒力基因等。该软件未来将面向宏基因组研究及未知物种快速自动化鉴定。
通过以上探索性工作,本课题组系统性地将纳米孔测序技术应用于病原菌的基因组组装分析,建立了基于纳米孔测序技术的细菌性传染病病原体分析模式。可以预见,Oxford Nanopore技术以其测序速度更快、超长读长、现场操作性更强等优势将为未来传染病疫情现场控制模式带来技术革命。
(中国疾控中心传染病所传染病诊断室
尤元海供稿/张建中研究员课题组)
参考文献:
[1] You Y, Davies MR, Protani M, McIntyre L, Walker MJ, Zhang J. Scarlet Fever Epidemic in China Caused by Streptococcus pyogenes Serotype M12: Epidemiologic and Molecular Analysis. EBioMedicine. 2018 Feb;28:128-135.
doi: 10.1016/j.ebiom.2018.
01.010. Epub 2018 Jan 11.
[2] You Y, Kou Y, Niu L, Jia Q, Liu Y, Davies MR, Walker MJ, Zhu J, Zhang J. 2018. Complete genome sequence of a Streptococcus pyogenes serotype M12 scarlet fever outbreak isolate from China, compiled using Oxford Nanopore and Illumina sequencing. Genome Announc 6:e00389-18.
https://doi.org/10.1128/genomeA.00389-18
Link: http://genomea.asm.org/content/6/18/e00389-18.full


